Wiki des méthodes de biologie moléculaire et cellulaire
Glossaire collaboratif des méthodes expérimentales de biologie moléculaire et cellulaire. Ce glossaire est réalisé par les étudiants du module "Méthodes expérimentales de biologie moléculaire et cellulaire" sous la supervision de l'équipe de Génétique Animale.
Spécial | A | B | C | D | E | F | G | H | I | J | K | L | M | N | O | P | Q | R | S | T | U | V | W | X | Y | Z | Tout
Q |
|---|
| EB | Quantification de m6A par RT-qPCR | ||
|---|---|---|---|
Quantification de m6A par RT-qPCR I) ObjectifsDe récentes études ont mis en avant l’importance de l’épigénétique dans les mécanismes de régulation des gènes. Parmi ces modifications, la N6-méthyladénosine (m6A) possède une place importante puisqu’il s’agit de la modification épigénétique la plus répandue sur les ARNm et ARNlnc des Eucaryotes (Jiang et al., 2021). Ce changement au niveau de l’ADN peut modifier la quantité de protéines traduite et peut avoir des conséquences diverses comme par exemple sur la consolidation de la mémoire et de la plasticité synaptique. La quantification de cette marque épigénétique est principalement réalisée par des techniques basées sur du séquençage ou de l’immunoprécipitation. Ces méthodes peuvent être difficiles et coûteuses à mettre en place, ou alors ne permettent que d’obtenir une résolution de l’ordre d’une centaine de nucléotides (Linder et al., 2015). La quantification de m6A par RT-qPCR permet de mesurer le niveau de méthylation d’une adénosine pour un coût plus faible et avec une meilleure résolution que les techniques existantes (Castellanos-Rubio et al., 2019). II) PrincipeCette quantification du niveau de méthylation des m6A est basée sur la différence d'efficacité de deux enzymes à rétrotranscrire l'ARN en ADNc Ces deux enzymes sont la MultiScribe™ Reverse Transcriptase (MRT) produite par Thermo Fisher Scientific (référence # 4311235) et la Bst DNA Polymerase, issue de Bacillus stearothermophilus, et produite par New England Biolabs (référence #M0275). La Bst a une capacité de rétrotranscription plus faible que l'enzyme MRT lorsque son amorce est fixée sur un nucléotide avant un site méthylé. Cela se traduit par un ralentissement de la vitesse de réaction de la Bst. Une plus petite quantité d'ADNc sera donc produite par la Bst comparée à la MRT. Ce ralentissement et donc cette différence de quantité d’ADNc pourra ainsi être quantifiée par qPCR, traduisant le niveau de méthylation de l’adénine ciblée (Figure 1).
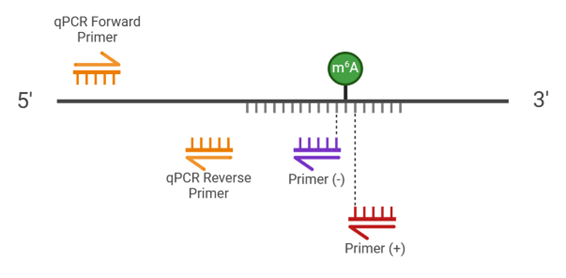
Figure 1 : Méthode de quantification du niveau relatif de m6A par RT-qPCR (Figure réalisée par Elio Bobe avec Biorender) III) Mode opératoireDans un premier temps, il faut concevoir deux amorces de RT-PCR, l'une juste avant (amorce (-)) et l'autre juste après (amorce (+)) le site potentiellement méthylé. Ces deux amorces ne doivent pas chevaucher ce site et en être séparées par un nucléotide (Figure 2). L’échantillon à analyser devra subir une RT-PCR avec chacune de ces deux amorces de manière indépendante, et ce pour chaque enzyme. Chacun de ces échantillons devra ensuite être analysé par qPCR en utilisant deux autres amorces situées en aval du site méthylé (Figure 2).
Figure 2 : Représentation graphique de la localisation des amorces (Figure réalisée par Elio Bobe avec Biorender) IV) Présentation des résultatsLes résultats sont analysés en utilisant la formule suivante :
Les résultats se présentent sous la forme de graphiques :
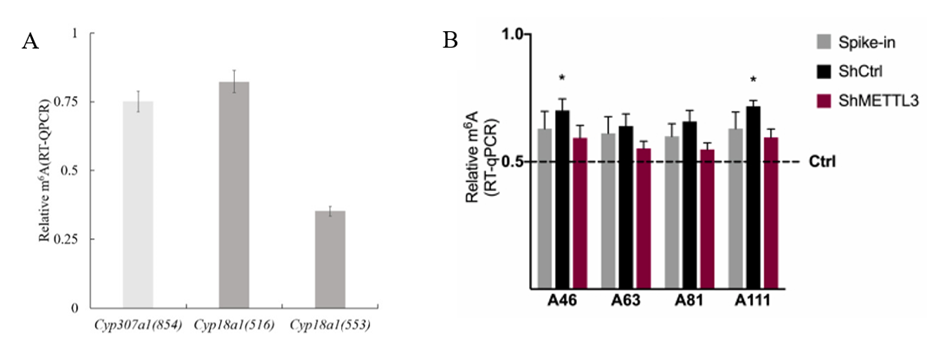
Figure 3 : Exemple de quantification de m6A par qPCR. (A) Relative quantification of the m6A levels of Cyp307a1 and Cyp18a1 in BmN cells (Chen et al., 2022). (B) m6A-qRT-PCR validation of Nanopore sequencing results (Pierattini et al., 2023). V) Interprétation des résultatsLes résultats ayant une valeur supérieure à 0,5 pourront être considérés comme méthylés, et ceux ayant une valeur inférieure seront considérés comme non-méthylés. Pour comparer des échantillons méthylés entre eux, il faudra effectuer différents tests statistiques afin de comparer les différents niveaux relatifs de méthylation. VI) Intérêts et limitesLes intérêts de cette méthode sont multiples. Elle présente un faible coût et sa facilité de mise en œuvre et d'analyse des résultats en font un outil pratique et efficace. La précision de la méthode est également un avantage notable, permettant une résolution d'un nucléotide. De plus, elle offre une quantification relative du niveau de méthylation réel des ARNm. Cependant, quelques limites sont à prendre en compte. Tout d'abord, la nécessité de savoir quelle adénine tester implique d'avoir accès au génome de l'échantillon. De plus, le design des amorces peut être difficile, surtout en présence de deux sites potentiellement méthylés à proximité, ce qui peut compliquer l'interprétation des résultats. VII) Références bibliographiquesCastellanos-Rubio A, Santin I, Olazagoitia-Garmendia A, Romero-Garmendia I, Jauregi-Miguel A, Legarda M, Bilbao JR (2019). A novel RT-QPCR-based assay for the relative quantification of residue specific m6A RNA methylation. Sci Rep 9: 4220 Chen Y, Fan B, Yasen A, Zhu J, Wang M, Shen X (2022). YTHDF3 Is Involved in the Diapause Process of Bivoltine Bombyx mori Strains by Regulating the Expression of Cyp307a1 and Cyp18a1 Genes in the Ecdysone Synthesis Pathway. Biomolecules 12: 1127 Jiang X, Liu B, Nie Z, Duan L, Xiong Q, Jin Z, Yang C, Chen Y (2021). The role of m6A modification in the biological functions and diseases. Signal Transduct Target Ther 6: 74 Linder B, Grozhik AV, Olarerin-George AO, Meydan C, Mason CE, Jaffrey SR (2015). Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nat Methods 12: 767–772 Pierattini B, D'Agostino S, Bon C, Peruzzo O, Alendar A, Codino A, Ros G, Persichetti F, Sanges R, Carninci P, Santoro C, Espinoza S, Valentini P, Pandolfini L, Gustincich S (2023). SINEUP non-coding RNA activity depends on specific N6-methyladenosine nucleotides. Mol Ther Nucleic Acids 32: 402–414
| |||